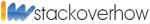Я создаю простой линейный график с помощью ggplot2 с пиками "сигнала" с течением времени по оси x.
Для небольшого пояснения: этот конкретный график — это то, что называется "хроматограммой", и показывает интенсивность сигнала в относительных единицах флуоресценции, нанесенную на график с течением времени. Основания ДНК (одно из A, C, G, T) "называются" (назначаются) при каждой интенсивности пика.
Я использую пример файла sangerseqR ab1 для моего MWE ниже. Я просто загружаю данные, создаю фрейм данных с данными трассировки для построения графика (значения интенсивности) и определяю последовательность ДНК как каждое из оснований, вызываемых на пиках, в определенные временные точки по оси x.
Я просто рисую небольшую часть последовательности ДНК (обрезанную спереди и сзади) для простоты.
Все отлично и работает так, как и ожидалось:
Это создает следующий график, который выглядит идеально (идентично выходным данным функции sangerseqR::chromatogram()), но не совсем такой, как мне нужно.
Обратите внимание на следующее. В идеальной хроматограмме мы должны видеть равномерно расположенные пики (и основания, вызываемые на пиках), но это бывает редко, и, конечно, не в этом примере.
Для моих целей (я хочу сравнить несколько почти идентичных последовательностей, выровняв/наложив их хроматограммы), мне нужно, чтобы разрывы оси x на пиках (соответствующие вызываемым основаниям) были равномерно расположены.
Я хочу игнорировать переменную времени и чтобы основания появлялись на равном расстоянии друг от друга. С технической точки зрения, "окна вызова оснований" должны иметь одинаковую ширину.
Для этого пики на графике должны быть соответственно расширены или сужены. Это то, что делает разное коммерческое программное обеспечение, но я не могу придумать, какую формулу применить к данным, чтобы я мог визуализировать график таким образом.
Буду очень признателен за любую помощь! Большое спасибо.
#read in data from sangerseqR example
seq_obj <- sangerseqR::readsangerseq(system.file("extdata", "heterozygous.ab1", package = "sangerseqR"))
#create data frame with trace data to plot
trace_df <- as.data.frame(seq_obj@traceMatrix) #columns for A, C, G, T
names(trace_df) <- c('A','C','G','T')
trace_df$time <- seq_len(nrow(trace_df))
trace_df <- as.data.frame(tidyr::pivot_longer(trace_df, -time, names_to = "base", values_to = "signal"))
#create data frame with base (letter) calls at the specific times (corresponding with trace peaks)
basecall <- unlist(strsplit(toString(seq_obj@primarySeq), ""))
basepos <- seq_obj@peakPosMatrix[,1] #first column for primary seq
base_df <- data.frame(call=basecall, time=basepos, callnum5=seq_along(basepos), callnum3=rev(seq_along(basepos)))
#join both data frames
trace_df <- dplyr::left_join(trace_df, base_df, by="time")
trace_df$call <- ifelse(trace_df$call==trace_df$base, trace_df$call, NA)
#trim data from 5' (top) and 3' (bottom) to plot only a small section of the sequence
trim5 <- 50
trim3 <- 500
startpos <- min(which(trace_df$callnum5==trim5))
endpos <- max(which(trace_df$callnum3==trim3))
trace_sub <- trace_df[startpos:endpos,]
#define colors, x-axis breaks and labels
basecolors <- c("green","blue","black","red")
xbreaks <- trace_sub$time[which(trace_sub$call==trace_sub$base)]
xlabels <- trace_sub$call[which(trace_sub$call==trace_sub$base)]
#make plot in ggplot2
P <- ggplot2::ggplot(trace_sub, ggplot2::aes(x=time, y=signal, group=base, colour=base)) +
ggplot2::geom_line(linewidth=0.5) +
ggplot2::scale_color_manual(values=basecolors) +
ggplot2::scale_x_continuous(breaks=xbreaks, labels=xlabels) +
ggplot2::theme_light()
grDevices::pdf(file="test.pdf", height=2, width=20)
print(P)
grDevices::dev.off()